Uso de modelos de regressão com Ridge e Lasso
Apresentação dos dados
Como continuação da Parte 2, nesse fase estaremos criando modelos de regressão, comparando-os e realizando predição de dados. Continuamos trabalhando com disciplinas dos dois primeiros semestres para a criação de um modelo que consiga calcular o cra de uma determinada matrícula. Para isso dividimos os dados em treino, validação e teste.
Legenda
- Cálculo.1: Cálculo Diferencial e Integral I - Período 1
- Vetorial: Álgebra Vetorial e Geometria Analítica - Período 1
- LPT: Leitura e Produção de Textos - Período 1
- P1: Programação I - Período 1
- LP1: Laboratório de Programação I - Período 1
- IC: Introdução à Computação - Período 1
- Cálculo.2: Cálculo Diferencial e Integral II - Período 2
- Discreta: Matemática Discreta - Período 2
- P2: Programação II - Período 2
- LP2: Laboratório de Programação II - Período 2
- Grafos: Teoria dos Grafos - Período 2
- Física.3: Fundamentos de Física Clássica - Período 2
dados_treino = read.csv("p1p2.graduados_treino.csv")
dados_validacao = read.csv("p1p2.graduados_validacao.csv")
dados_teste = read.csv("test.csv")
colnames(dados_teste) = c("matricula", "Cálculo.1", "Vetorial", "LPT", "P1", "LP1", "IC", "Cálculo.2", "Discreta", "P2", "LP2", "Grafos", "Física.3")
dados_treino = dados_treino[,2:15]
dados_validacao = dados_validacao[,2:15]
head(dados_treino)## Matricula Cálculo.1 Vetorial LPT P1 LP1 IC
## 1 002b348d3bc88b68aa6ebd24cfc9d6a0 5.6 7.5 9.1 8.0 8.7 8.4
## 2 007961bf3929ae353c8585e6c09df369 6.7 8.5 8.6 7.1 7.6 7.7
## 3 00960ff73dd29b59f4432072c2219c04 7.0 5.6 6.5 7.3 8.5 7.5
## 4 00b6f55e3639c9d3b835751a39ca6309 NA 7.3 NA NA NA NA
## 5 00eb58fa89c562efda118e64efab77a5 5.5 7.0 7.4 6.8 6.3 8.6
## 6 01fc383ae2c2cb3e69d844272a5ecac0 6.6 5.9 8.3 9.1 8.8 9.5
## Cálculo.2 Discreta P2 LP2 Grafos Física.3 cra
## 1 NA 9.2 7.7 8.6 7.2 9.6 8.109877
## 2 NA 5.0 5.7 8.0 7.0 7.8 6.975824
## 3 NA 5.3 5.6 8.1 7.9 7.0 6.057282
## 4 7.2 NA NA NA NA NA 7.766667
## 5 NA 6.7 7.9 9.0 6.1 7.1 7.693827
## 6 NA 6.1 6.8 9.0 5.3 7.3 6.722892Percebemos que algumas notas de disciplinas estão com valores vazios. Para que não precisemos remover toda a linha do data frame, vamos realizar imputação de dados. Decidimos que os valores vazios em disciplinas serão substituídos pelo valor do cra de suas respectivas matrículas.
for(i in 1:nrow(dados_treino)){
for(j in 1:ncol(dados_treino)){
if(is.na(dados_treino[i,j])){
dados_treino[i, j] = dados_treino$cra[i]
}
if(i <= nrow(dados_validacao)){
if(is.na(dados_validacao[i,j])){
dados_validacao[i, j] = dados_validacao$cra[i]
}
}
}
}
head(dados_treino)## Matricula Cálculo.1 Vetorial LPT
## 1 002b348d3bc88b68aa6ebd24cfc9d6a0 5.600000 7.5 9.100000
## 2 007961bf3929ae353c8585e6c09df369 6.700000 8.5 8.600000
## 3 00960ff73dd29b59f4432072c2219c04 7.000000 5.6 6.500000
## 4 00b6f55e3639c9d3b835751a39ca6309 7.766667 7.3 7.766667
## 5 00eb58fa89c562efda118e64efab77a5 5.500000 7.0 7.400000
## 6 01fc383ae2c2cb3e69d844272a5ecac0 6.600000 5.9 8.300000
## P1 LP1 IC Cálculo.2 Discreta P2 LP2
## 1 8.000000 8.700000 8.400000 8.109877 9.200000 7.700000 8.600000
## 2 7.100000 7.600000 7.700000 6.975824 5.000000 5.700000 8.000000
## 3 7.300000 8.500000 7.500000 6.057282 5.300000 5.600000 8.100000
## 4 7.766667 7.766667 7.766667 7.200000 7.766667 7.766667 7.766667
## 5 6.800000 6.300000 8.600000 7.693827 6.700000 7.900000 9.000000
## 6 9.100000 8.800000 9.500000 6.722892 6.100000 6.800000 9.000000
## Grafos Física.3 cra
## 1 7.200000 9.600000 8.109877
## 2 7.000000 7.800000 6.975824
## 3 7.900000 7.000000 6.057282
## 4 7.766667 7.766667 7.766667
## 5 6.100000 7.100000 7.693827
## 6 5.300000 7.300000 6.722892Como resultado final e para uma apresentação dos dados antes a criação de modelos, podemos ver abaixo a tabela de correlação entre a variáveis.
M = cor(na.omit(dados_treino[,2:14]))
corrplot(M, type = "lower", title = "Correlação de disciplinas", order="hclust", col=brewer.pal(n=7, name="PuOr"), addCoef.col = "black", tl.col="black", tl.srt=45, mar=c(0,0,1,0) )
Modelo de regressão Ridge
Iniciaremos utilizando o método de regularização Ridge Regression, que suaviza atributos relacionados e que aumentam o ruído no modelo. Porque queremos encontrar o bom valor lambda para o modelo, utilizamos nos dados de treino a função train para escolher aquele com menor RMSE dentre 100 possíveis valores lambda.
set.seed(825)
fitControl <- trainControl(method = "cv",
number = 10)
lambdaGrid <- expand.grid(lambda = 10^seq(10, -2, length=100))
ridge <- train(cra~., data = dados_treino[,2:14],
method='ridge',
trControl = fitControl,
tuneGrid = lambdaGrid,
preProcess=c('center', 'scale')
)Abaixo vemos a sequência de valores obtidos e uma representação gráfica do mesmo.
ridge## Ridge Regression
##
## 511 samples
## 12 predictor
##
## Pre-processing: centered (12), scaled (12)
## Resampling: Cross-Validated (10 fold)
## Summary of sample sizes: 460, 461, 461, 460, 459, 459, ...
## Resampling results across tuning parameters:
##
## lambda RMSE Rsquared MAE
## 1.000000e-02 0.4666691 0.7205067 0.3533620
## 1.321941e-02 0.4666576 0.7205613 0.3533882
## 1.747528e-02 0.4666553 0.7206293 0.3534281
## 2.310130e-02 0.4666746 0.7207123 0.3535139
## 3.053856e-02 0.4667386 0.7208104 0.3536408
## 4.037017e-02 0.4668891 0.7209215 0.3538404
## 5.336699e-02 0.4672012 0.7210388 0.3541924
## 7.054802e-02 0.4678056 0.7211484 0.3548253
## 9.326033e-02 0.4689280 0.7212263 0.3559141
## 1.232847e-01 0.4709501 0.7212350 0.3577424
## 1.629751e-01 0.4745053 0.7211219 0.3609009
## 2.154435e-01 0.4806172 0.7208214 0.3654819
## 2.848036e-01 0.4908814 0.7202617 0.3732596
## 3.764936e-01 0.5076614 0.7193782 0.3861417
## 4.977024e-01 0.5342141 0.7181299 0.4082041
## 6.579332e-01 0.5746033 0.7165155 0.4426444
## 8.697490e-01 0.6332700 0.7145812 0.4938157
## 1.149757e+00 0.7142690 0.7124167 0.5632882
## 1.519911e+00 0.8203771 0.7101393 0.6530969
## 2.009233e+00 0.9523370 0.7078718 0.7608911
## 2.656088e+00 1.1084045 0.7057216 0.8867547
## 3.511192e+00 1.2842648 0.7037675 1.0263318
## 4.641589e+00 1.4733720 0.7020545 1.1752311
## 6.135907e+00 1.6677309 0.7005976 1.3294585
## 8.111308e+00 1.8590149 0.6993891 1.4822005
## 1.072267e+01 2.0397497 0.6984067 1.6266907
## 1.417474e+01 2.2042385 0.6976208 1.7575687
## 1.873817e+01 2.3490101 0.6970001 1.8725307
## 2.477076e+01 2.4727655 0.6965148 1.9708844
## 3.274549e+01 2.5759678 0.6961382 2.0530252
## 4.328761e+01 2.6602781 0.6958478 2.1201178
## 5.722368e+01 2.7280095 0.6956248 2.1739676
## 7.564633e+01 2.7816953 0.6954543 2.2166214
## 1.000000e+02 2.8237972 0.6953241 2.2500547
## 1.321941e+02 2.8565400 0.6952251 2.2760463
## 1.747528e+02 2.8818396 0.6951498 2.2961308
## 2.310130e+02 2.9012901 0.6950926 2.3115730
## 3.053856e+02 2.9161862 0.6950492 2.3233975
## 4.037017e+02 2.9275607 0.6950163 2.3324254
## 5.336699e+02 2.9362266 0.6949914 2.3393029
## 7.054802e+02 2.9428176 0.6949725 2.3445334
## 9.326033e+02 2.9478239 0.6949582 2.3485060
## 1.232847e+03 2.9516228 0.6949474 2.3515205
## 1.629751e+03 2.9545032 0.6949392 2.3538060
## 2.154435e+03 2.9566861 0.6949330 2.3555381
## 2.848036e+03 2.9583396 0.6949283 2.3568503
## 3.764936e+03 2.9595916 0.6949248 2.3578448
## 4.977024e+03 2.9605395 0.6949221 2.3585977
## 6.579332e+03 2.9612570 0.6949201 2.3591675
## 8.697490e+03 2.9617999 0.6949185 2.3595988
## 1.149757e+04 2.9622108 0.6949174 2.3599251
## 1.519911e+04 2.9625217 0.6949165 2.3601721
## 2.009233e+04 2.9627569 0.6949158 2.3603589
## 2.656088e+04 2.9629349 0.6949153 2.3605002
## 3.511192e+04 2.9630695 0.6949149 2.3606072
## 4.641589e+04 2.9631714 0.6949147 2.3606881
## 6.135907e+04 2.9632484 0.6949144 2.3607493
## 8.111308e+04 2.9633067 0.6949143 2.3607956
## 1.072267e+05 2.9633508 0.6949141 2.3608306
## 1.417474e+05 2.9633842 0.6949141 2.3608571
## 1.873817e+05 2.9634094 0.6949140 2.3608771
## 2.477076e+05 2.9634285 0.6949139 2.3608923
## 3.274549e+05 2.9634429 0.6949139 2.3609038
## 4.328761e+05 2.9634539 0.6949139 2.3609125
## 5.722368e+05 2.9634621 0.6949138 2.3609190
## 7.564633e+05 2.9634684 0.6949138 2.3609240
## 1.000000e+06 2.9634731 0.6949138 2.3609277
## 1.321941e+06 2.9634767 0.6949138 2.3609306
## 1.747528e+06 2.9634794 0.6949138 2.3609327
## 2.310130e+06 2.9634814 0.6949138 2.3609344
## 3.053856e+06 2.9634830 0.6949138 2.3609356
## 4.037017e+06 2.9634842 0.6949138 2.3609365
## 5.336699e+06 2.9634851 0.6949138 2.3609372
## 7.054802e+06 2.9634857 0.6949138 2.3609378
## 9.326033e+06 2.9634862 0.6949138 2.3609382
## 1.232847e+07 2.9634866 0.6949138 2.3609385
## 1.629751e+07 2.9634869 0.6949138 2.3609387
## 2.154435e+07 2.9634871 0.6949138 2.3609389
## 2.848036e+07 2.9634873 0.6949138 2.3609390
## 3.764936e+07 2.9634874 0.6949138 2.3609391
## 4.977024e+07 2.9634875 0.6949138 2.3609392
## 6.579332e+07 2.9634876 0.6949138 2.3609392
## 8.697490e+07 2.9634876 0.6949138 2.3609393
## 1.149757e+08 2.9634877 0.6949138 2.3609393
## 1.519911e+08 2.9634877 0.6949138 2.3609393
## 2.009233e+08 2.9634877 0.6949138 2.3609394
## 2.656088e+08 2.9634877 0.6949138 2.3609394
## 3.511192e+08 2.9634878 0.6949138 2.3609394
## 4.641589e+08 2.9634878 0.6949138 2.3609394
## 6.135907e+08 2.9634878 0.6949138 2.3609394
## 8.111308e+08 2.9634878 0.6949138 2.3609394
## 1.072267e+09 2.9634878 0.6949138 2.3609394
## 1.417474e+09 2.9634878 0.6949138 2.3609394
## 1.873817e+09 2.9634878 0.6949138 2.3609394
## 2.477076e+09 2.9634878 0.6949138 2.3609394
## 3.274549e+09 2.9634878 0.6949138 2.3609394
## 4.328761e+09 2.9634878 0.6949138 2.3609394
## 5.722368e+09 2.9634878 0.6949138 2.3609394
## 7.564633e+09 2.9634878 0.6949138 2.3609394
## 1.000000e+10 2.9634878 0.6949138 2.3609394
##
## RMSE was used to select the optimal model using the smallest value.
## The final value used for the model was lambda = 0.01747528.plot(ridge)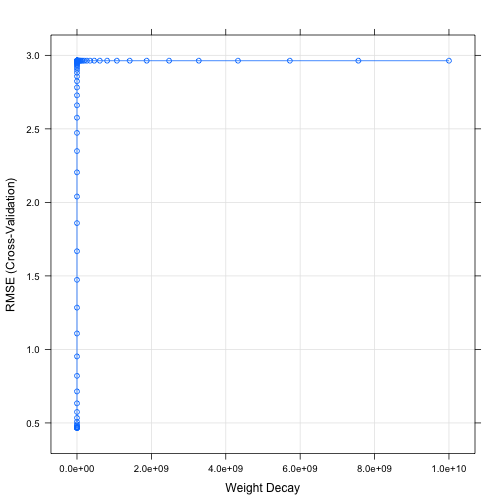
Em seguida, realizamos a predição dos dados, mas agora utilizando os dados de validação. Abaixo temos um gráfico representando a validação do modelo, incluindo a “linha preditora” e resíduos.
ridge.pred <- predict(ridge, dados_validacao[,2:14])
ridge.pred <- data.frame(pred = ridge.pred, obs = dados_validacao$cra)
ggplot(ridge.pred, aes(x = pred, y = obs)) + geom_point(alpha = 0.5, position = position_jitter(width=0.2)) + geom_abline(colour = "darkred") + ggtitle("Previsão x Observado (validação)")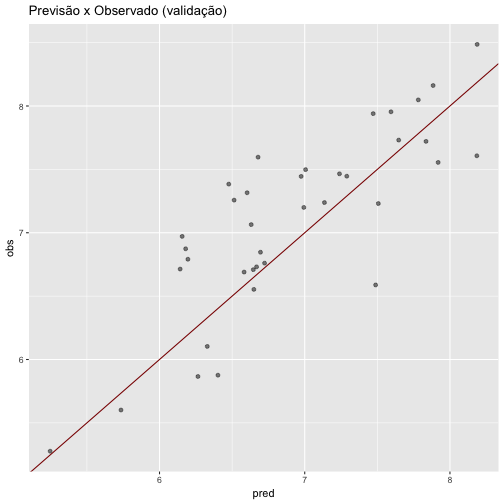
O RMSE alto indica que o erro na predição foi alto. Ao utlizar a função round é fornecido que o RMSE obtido no modelo Ridge foi igual a 0.449.
round(defaultSummary(ridge.pred), digits = 3)## RMSE Rsquared MAE
## 0.450 0.667 0.378Modelo de regressão Lasso
Assim como no tópico anterior, criaremos um novo modelo agora utilizando o método Lasso para a seleção de preditores. Após a criação e treinamento do modelo, também realizaremos a predição do mesmo.
lasso <- train(cra~., data = dados_treino[,2:14],
method='lasso',
tuneLength = 10,
preProcess=c('center', 'scale')
)O gráfico abaixo ilustra os 10 valores obtidos para fraction e a relação com seus respectivos RMSE. Em seguida os mesmos valores em detalhes.
plot(lasso)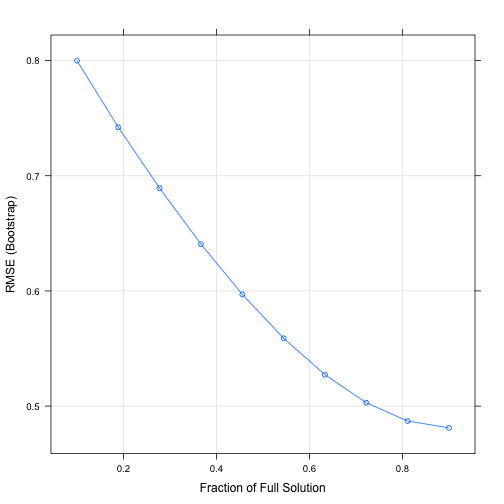
lasso## The lasso
##
## 511 samples
## 12 predictor
##
## Pre-processing: centered (12), scaled (12)
## Resampling: Bootstrapped (25 reps)
## Summary of sample sizes: 511, 511, 511, 511, 511, 511, ...
## Resampling results across tuning parameters:
##
## fraction RMSE Rsquared MAE
## 0.1000000 0.7998468 0.4808770 0.6355545
## 0.1888889 0.7420451 0.5357436 0.5889774
## 0.2777778 0.6891438 0.5912402 0.5451351
## 0.3666667 0.6406054 0.6288635 0.5039799
## 0.4555556 0.5970528 0.6547851 0.4666116
## 0.5444444 0.5589272 0.6726206 0.4332034
## 0.6333333 0.5272892 0.6846359 0.4048229
## 0.7222222 0.5029228 0.6929940 0.3818566
## 0.8111111 0.4870511 0.6985747 0.3649329
## 0.9000000 0.4811501 0.7005923 0.3577942
##
## RMSE was used to select the optimal model using the smallest value.
## The final value used for the model was fraction = 0.9.Para realizar a predição dos dados também utilizamos os dados de validação. Abaixo temos um gráfico representando a validação do modelo, incluindo a “linha preditora” e resíduos.
lasso.pred <- predict(lasso, dados_validacao[,2:14])
lasso.pred <- data.frame(pred = lasso.pred, obs = dados_validacao$cra)
ggplot(lasso.pred, aes(x = pred, y = obs)) + geom_point(alpha = 0.5, position = position_jitter(width=0.2)) + geom_abline(colour = "darkred") + ggtitle("Previsão x Observado (validação)")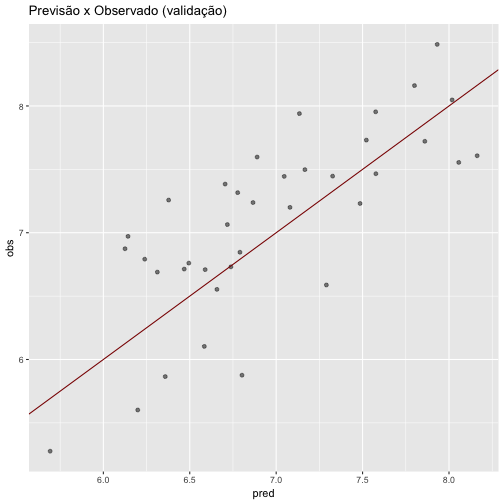
Utilizando a função round é fornecido que o RMSE obtido no modelo Ridge foi igual a 0.437.
round(defaultSummary(lasso.pred), digits = 3)## RMSE Rsquared MAE
## 0.437 0.663 0.373Comparação de modelos
Realizamos a comparação de modelos com base nos valores obtidos no RMSE. Quanto mais baixo o valor, melhor. Abaixo temos os gráficos mostrados anteriormente, agora lado a lado para uma melhor comparação.Vemos que o comportamento nos dados de validação é bem semelhante, os gráficos gerados não apresentam uma diferença perceptível muito significante.
compare <- ridge.pred
compare$model <- "Ridge"
lasso.pred$model <- "Lasso"
compare <- rbind(compare, lasso.pred)
ggplot(compare, aes(x = pred, y = obs)) +
geom_point(alpha = 0.5, position = position_jitter(width=0.2)) +
facet_grid(. ~ model) +
geom_abline() +
ggtitle("Observado x Previsão (validação)")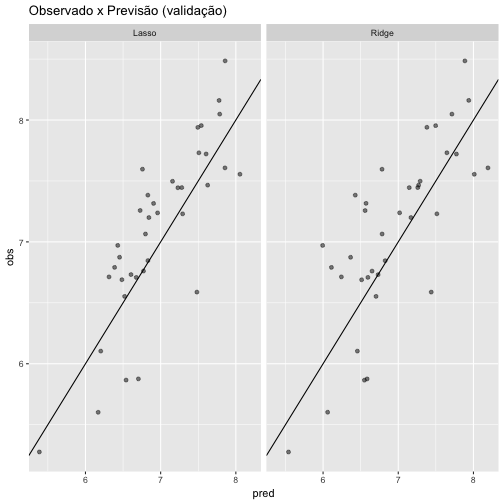
Assim como nos gráficos, o fenômeno se repetiu para os valores de RSME. Ambos são altos e com quase o mesmo valor. Porém, o RMSE do modelo Lasso consegue atingir um menor valor. Podemos então considerar que o melhor modelo é o que utiliza da técnica Lasso. O melhor modelo é aquele que possui o RMSE mais baixo.
round(defaultSummary(ridge.pred), digits = 3)## RMSE Rsquared MAE
## 0.450 0.667 0.378round(defaultSummary(lasso.pred), digits = 3)## RMSE Rsquared MAE
## 0.437 0.663 0.373Importância de variáveis no modelo Lasso
Geramos uma representação gráfica com a importância das variáveis e mais abaixo estão os valores detalhados sobre a importância das mesmas. Vemos que Cálculo 2 é apresentada como a variável de maior importância, seguida por IC e P2. Temos apenas uma variável discartada que foi Física 3, apresentando overall igual a 0.
plot(varImp(lasso))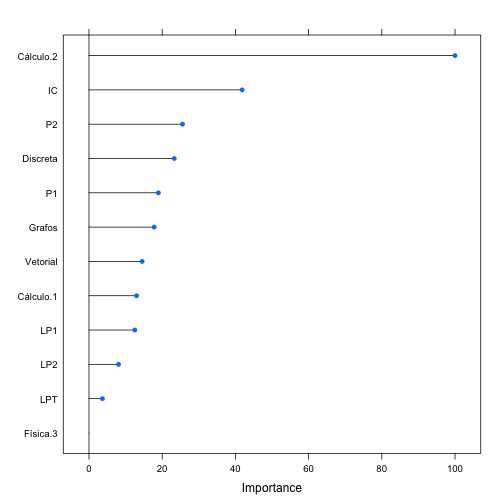
varImp(lasso)## loess r-squared variable importance
##
## Overall
## Cálculo.2 100.000
## IC 41.837
## P2 25.549
## Discreta 23.305
## P1 18.968
## Grafos 17.809
## Vetorial 14.513
## Cálculo.1 13.001
## LP1 12.518
## LP2 8.069
## LPT 3.667
## Física.3 0.000Re-treino de modelo Lasso com dados de validação
Repetiremos aqui os passos realizados na construção e treino do modelo Lasso. Porém, desta vez estaremos utilizando os dados de validação para treinar o modelo. Segue:
lasso <- train(cra~., data = dados_validacao[,2:14],
method='lasso',
tuneLength = 10,
preProcess=c('center', 'scale'))O gráfico abaixo ilustra os 10 valores obtidos para fraction e a relação com seus respectivos RMSE. Em seguida os mesmos valores em detalhes.
plot(lasso)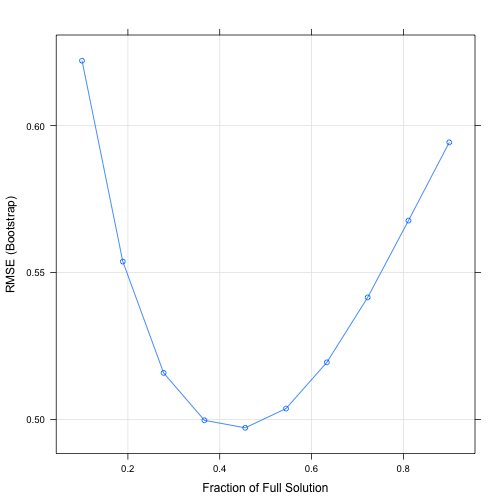
lasso## The lasso
##
## 37 samples
## 12 predictors
##
## Pre-processing: centered (12), scaled (12)
## Resampling: Bootstrapped (25 reps)
## Summary of sample sizes: 37, 37, 37, 37, 37, 37, ...
## Resampling results across tuning parameters:
##
## fraction RMSE Rsquared MAE
## 0.1000000 0.6221030 0.6100633 0.4834311
## 0.1888889 0.5537367 0.5910030 0.4262028
## 0.2777778 0.5158326 0.5758529 0.4060596
## 0.3666667 0.4996999 0.5693654 0.4061517
## 0.4555556 0.4971366 0.5642472 0.4115263
## 0.5444444 0.5037047 0.5587759 0.4191394
## 0.6333333 0.5194272 0.5493267 0.4324576
## 0.7222222 0.5415312 0.5363950 0.4502765
## 0.8111111 0.5676876 0.5190685 0.4702847
## 0.9000000 0.5943074 0.4995609 0.4909675
##
## RMSE was used to select the optimal model using the smallest value.
## The final value used for the model was fraction = 0.4555556.Em seguida, a realização de predição utilizando os mesmos dados. Abaixo temos um gráfico representando a validação do modelo, incluindo a “linha preditora” e resíduos.
lasso.pred <- predict(lasso, dados_validacao[,2:14])
lasso.pred <- data.frame(pred = lasso.pred, obs = dados_validacao$cra)
ggplot(lasso.pred, aes(x = pred, y = obs)) + geom_point(alpha = 0.5, position = position_jitter(width=0.2)) + geom_abline(colour = "darkred") + ggtitle("Previsão x Observado (validação)")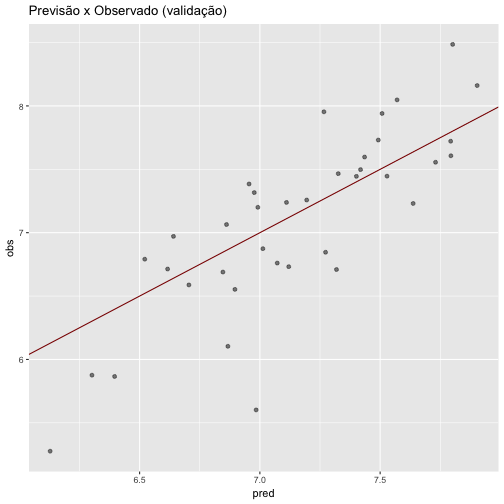
Utilizando a função round é fornecido que o RMSE obtido no modelo Ridge foi igual a 0.454. Ao interpretar o resultado da função entendemos que houve um aumento no valor do RMSE e, consequentemente, uma piora no modelo.
round(defaultSummary(lasso.pred), digits = 3)## RMSE Rsquared MAE
## 0.416 0.718 0.327Tentativa de melhora no modelo
Para tentar melhorar o modelo, vamos tentar uma nova abordagem para imputação de dados. Inicialmente, fazíamos a substituição de valores NA pelo valor do cra. Agora substituiremos esses valores vazios pela média das notas do primeiro período.
dados_treino = read.csv("p1p2.graduados_treino.csv")
dados_validacao = read.csv("p1p2.graduados_validacao.csv")
dados_treino = dados_treino[,2:15]
dados_validacao = dados_validacao[,2:15]
for(i in 1:nrow(dados_treino)){
for(j in 1:ncol(dados_treino)){
if(is.na(dados_treino[i,j])){
dados_treino[i, j] = rowMeans(dados_treino[i,2:13], na.rm = T)
}
if(i <= nrow(dados_validacao)){
if(is.na(dados_validacao[i,j])){
dados_validacao[i, j] = rowMeans(dados_treino[i,2:13], na.rm = T)
}
}
}
}Assim como nos tópicos anteriores, criamos o modelo utlizando os dados de treino.
lasso <- train(cra~., data = dados_treino[,2:14],
method='lasso',
tuneLength = 10,
preProcess=c('center', 'scale'))O gráfico abaixo ilustra os 10 valores obtidos para fraction e a relação com seus respectivos RMSE. Em seguida os mesmos valores em detalhes.
plot(lasso)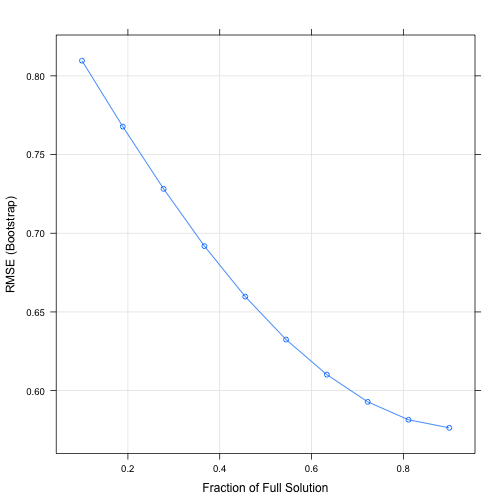
lasso## The lasso
##
## 511 samples
## 12 predictor
##
## Pre-processing: centered (12), scaled (12)
## Resampling: Bootstrapped (25 reps)
## Summary of sample sizes: 511, 511, 511, 511, 511, 511, ...
## Resampling results across tuning parameters:
##
## fraction RMSE Rsquared MAE
## 0.1000000 0.8096196 0.3734452 0.6392078
## 0.1888889 0.7677505 0.4348288 0.6037849
## 0.2777778 0.7282028 0.4739862 0.5707756
## 0.3666667 0.6918218 0.4994136 0.5405741
## 0.4555556 0.6597383 0.5162044 0.5132659
## 0.5444444 0.6324679 0.5286369 0.4891104
## 0.6333333 0.6101479 0.5380087 0.4682531
## 0.7222222 0.5928893 0.5457078 0.4510502
## 0.8111111 0.5814896 0.5509442 0.4381402
## 0.9000000 0.5763620 0.5537305 0.4309036
##
## RMSE was used to select the optimal model using the smallest value.
## The final value used for the model was fraction = 0.9.Em seguida, a realização de predição utilizando os dados de validação. Abaixo temos um gráfico representando a validação do modelo, incluindo a “linha preditora” e resíduos.
lasso.pred <- predict(lasso, dados_validacao[,2:14])
lasso.pred <- data.frame(pred = lasso.pred, obs = dados_validacao$cra)
ggplot(lasso.pred, aes(x = pred, y = obs)) + geom_point(alpha = 0.5, position = position_jitter(width=0.2)) + geom_abline(colour = "darkred") + ggtitle("Previsão x Observado (validação)")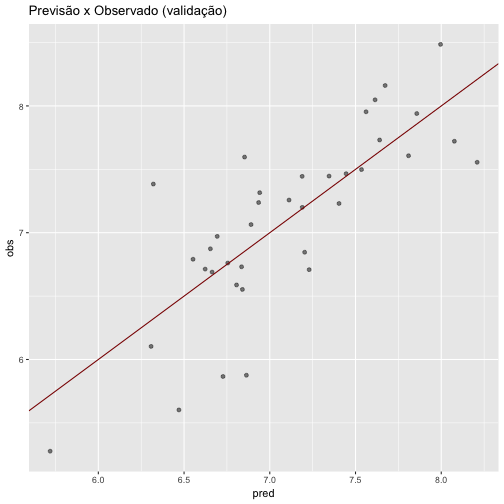
Geramos uma representação gráfica com a importância das variáveis e mais abaixo estão os valores detalhados sobre a importância das mesmas. Vemos que Cálculo 2 continua sendo apresentada como a variável de maior importância, seguida por IC e agora Grafos. Temos apenas uma variável discartada que para o novo modelo foi LPT, apresentando overall igual a 0.
plot(varImp(lasso))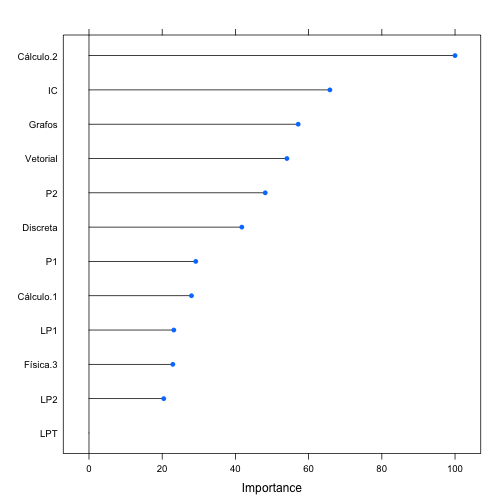
varImp(lasso)## loess r-squared variable importance
##
## Overall
## Cálculo.2 100.00
## IC 65.81
## Grafos 57.15
## Vetorial 54.07
## P2 48.15
## Discreta 41.75
## P1 29.17
## Cálculo.1 28.00
## LP1 23.17
## Física.3 22.91
## LP2 20.43
## LPT 0.00Utilizando a função round é fornecido que o RMSE obtido no modelo Ridge foi igual a 0.411. Ao interpretar o resultado da função entendemos que houve uma diminuição no valor do RMSE e, consequentemente, uma melhora no modelo.
round(defaultSummary(lasso.pred), digits = 3)## RMSE Rsquared MAE
## 0.411 0.670 0.343Gerando dados de predição
Para gerar dados de predição utilizamos dados de teste. Realizamos a predição de cra a partir das disciplinas dos dois primeiros semestres. O resultado foi submetido na plataforma Kaggle.
head(dados_teste)## matricula Cálculo.1 Vetorial LPT P1 LP1
## 1 037119c4740aa04d14742993e4aab789 8.2 9.1 8.3 7.0 10.0
## 2 0402a824ba0c87f73209cdb49b5072be 5.0 7.7 7.8 8.8 7.1
## 3 075d778a8444ef224757557479320148 5.2 5.0 9.6 7.8 6.2
## 4 09b9a1b335be033c236add2ba699c13f 7.6 6.0 8.7 7.0 8.4
## 5 0d3c0ddcdd2a97627575c27b5d1a058a 8.3 8.1 7.0 8.4 9.4
## 6 107f352e7c2d881c0f5f72a4584cd8fd 5.5 6.9 7.3 7.0 9.1
## IC Cálculo.2 Discreta P2 LP2 Grafos Física.3
## 1 7.7 9.2 9.7 9.2 8.9 9.1 9.0
## 2 8.8 7.3 7.3 7.1 7.3 8.0 6.0
## 3 5.4 5.1 5.0 5.7 7.0 7.0 9.3
## 4 7.0 5.0 5.9 7.8 6.0 6.6 5.9
## 5 8.4 7.8 7.9 8.5 8.5 8.0 8.0
## 6 7.4 7.0 7.6 9.4 5.2 8.4 8.8dados_teste$cra = NA
dados_teste$cra = predict(lasso, dados_teste)
dados_teste = dados_teste[,c("matricula","cra")]
head(dados_teste)## matricula cra
## 1 037119c4740aa04d14742993e4aab789 8.309662
## 2 0402a824ba0c87f73209cdb49b5072be 7.380805
## 3 075d778a8444ef224757557479320148 6.478184
## 4 09b9a1b335be033c236add2ba699c13f 6.809691
## 5 0d3c0ddcdd2a97627575c27b5d1a058a 7.816722
## 6 107f352e7c2d881c0f5f72a4584cd8fd 7.552772